Výzkum

Na této stránce bych rád shrnul a zpřístupnil výsledky svého zhruba 6 letého působení v Centru pro analýzu biomedicínského obrazu (CBIA) na Fakultě informatiky Masarykovy univerzity v Brně. CBIA je interdisciplinární skupina, sestávající z biologů, fyziků, matematiků a odborníků na výpočetní techniku. Jejím primárním zaměřením je pořizování 2D a 3D obrazů buněk a buněčných struktur na moderních fluorescenčních mikroskopech a jejich následné zpracování. Cílem těchto pozorování jsou snahy o porozumění organizaci a funkci buněčného materiálu a následné klinické aplikace zaměřené především na včasnou diagnózu a prevenci nádorových onemocnění.
Software
Jako zaměstnanec jsem se velkou měrou podílel zejména na vývoji následujícího software:
- Acquiarium (screenshoty) – Software pro automatizované snímání velkého množství 2D a 3D dat na fluorescenčních mikroskopech a jejich následné dávkové vyhodnocování (odstranění optických vad, segmentace, kolokalizace, statistiky, atd.). Jedná se o multi-platformní aplikaci vyvíjenou v C++ a wxWidgets. Osobně jsem se podílel zejména na zobrazovací a vyhodnocovací části software (mimojiné návrh a implementace dynamického systému pluginů).
- v3dlib – Multiplatformní knihovna naprogramovaná v C++ a wxWidgets. Určená k vizualizaci 2D a 3D dat z optických mikroskopů.
- i3dlib – Knihovna obsahující implementace mnoha záladních i pokročilých algoritmů z oblasti zpracování obrazu.
Výzkumná činnost

V rámci postgraduálního studia jsem se zabýval návrhem nových algoritmů pro rychlé a spolehlivé vyhodnocení 2D a 3D biomedicínských dat z fluorescenčních mikroskopů. Konkrétním tématem mé práce byla segmentace, čili detekce objektů (v tomto případě buněk a buněčných jader) nebo jejich hranic v daném obraze. Ideální segmentační algoritmus by měl být zcela automatický, přesný a dostatečně rychlý, aby bylo možné spolehlivě zpracovat velké množství dat bez zásahu uživatele. Z důvodu nízké kvality nasnímaných dat je však tento cíl pouze obtížně dosažitelný. Moje práce v této oblasti spadala pod grant MŠMT ČR nazvaný „Vytipování markerů, screening a včasná diagnostika nádorových onemocnění pomocí vysoce automatizovaného zpracování multidimenzionálních biomedicínských obrazů“. Ve velké míře jsem spolupracoval také s výzkumným střediskem Centro de investigación médica aplicada (CIMA) při Universidad de Navarra v Pamploně ve Španělsku, kde jsem dvě čtvrtletní stáže v letech 2008 a 2010.
Disertační práce a publikace
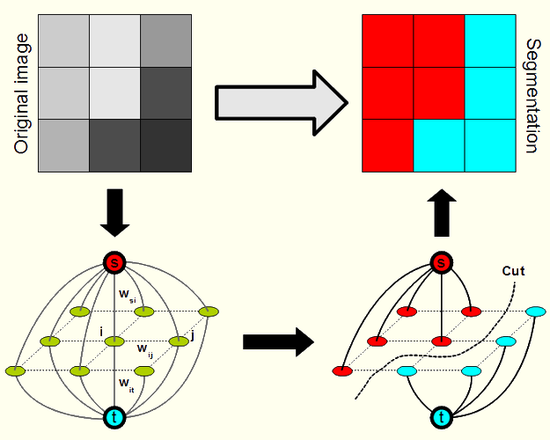
Ve svém studiu jsem využíval a vylepšoval přístupy, kde optimální segmentace je dána jako minimum určité vhodně zvolené energetické funkce. Pro nalezení tohoto minima lze použít různé matematické přístupy. V mém případě se jednalo o metody založené na kombinatorické optimalizaci, konkrétně na hledání minimálního řezu v grafu. Tento přístup vychází zejména z práce Y. Boykova (viz jeho vědecké publikace). Výsledky tohoto studia (viz níže) jsou přístupné ve formě mé disertační práce, volně šiřitelné knihovny a publikací prezentovaných v mezinárodních časopisech a konferencích:
Ilustrace metody:
Graph cut knihovna
Implementace většiny algoritmů vyvinutých a popsaných v disertační práci je volně přístupná v níže uvedené knihovně. Knihovna je naprogramována v C++ a je šířena pod GPL licencí.
- Stránka knihovny na webu CBIA s příklady použití
- Dokumentace
- Zdrojové kódy: knihovna (ZIP), rozhraní do MATLABu (ZIP)
Výsledky
Níže jsou popsáný a ilustrováný klíčové výsledky z mé disertační práce.
Aproximace metrik
Často používané energetické funkce obsahují členy závislé na délce hranice segmentace (hledáme segmentaci s krátkou, tj. hladkou, hranicí). Proto je třeba zabudovat do grafu aproximaci Euklidovské případně Riemannovské metriky. Pro data z fluorescenčních mikroskopů bylo třeba tyto aproximace přizpůsobit pro anisotropní rozlišení (různé rozlišení v různých osách). Součástí práce bylo i snížení chyby aproximace.
Aproximace Riemannovské metriky pro různě velká okolí (N4, N8, N6 a N32):




Rekonstrukce catenoidu pro různě velká okolí (N26, N98) a porovnání se spojitou rekonstrukcí:
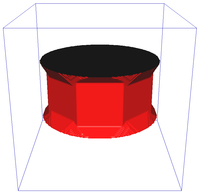
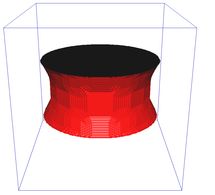
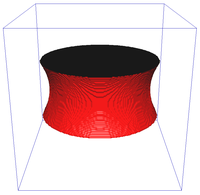
Segmentace obrazu
Pro segmentaci buňek a buněčných jader byly navrženy dva algoritmy využívající výše zmíněné aproximaci. První algoritmus je modifikací Chan-Vese algoritmu s vylepšenou inicializací, hladkostí hranice a nižšími paměťovými nároky:
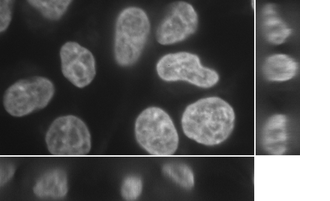

Algoritmus umožňuje i víceúrovňovou (na obrázku níže se jedná konkrétně 3 úrovně – pozadí, cytoplazma, jádro) segmentaci:
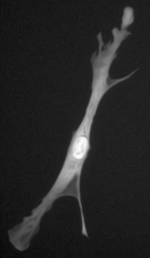
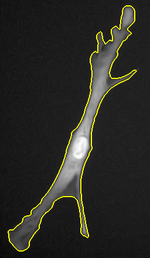
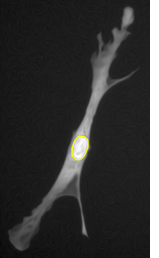

Druhý algoritmus je založený na geodesické segmentaci a je určený pro segmentaci clusterů dotýkajících se buněčných jader:
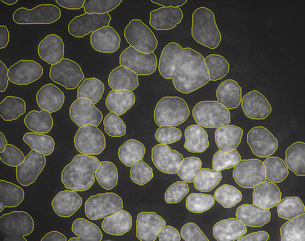
Zachovávání topologie a sledování pohybu
V závěrečné části práce jsou grafové algoritmy rozšířeny o zachovávání topologie, což umožňuje předem definovat počet hledaných objektů a jejich topologii. Příkladem je segmentace dvou označených kostí v MRI obrázku kolena:
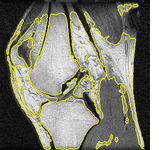
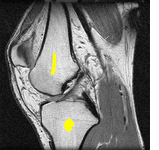
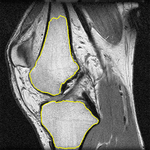
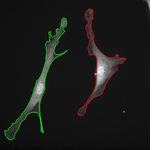
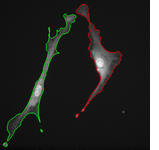
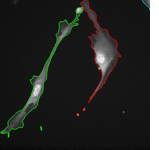
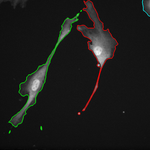
Topologická omezení lze rovněž kombinovat s Chan-Vese segmentací a využít je pro vytvoření metody na monitorování pohybu živých buněk v obrazové sekvenci, kde se jednotlivé objekty mohou dotýkat nebo dělit na více částí: